पत्ता लगाउने विशिष्टता
धेरैजसो अवस्थामा, प्राइमर डिजाइनको उद्देश्य पीसीआरको विशिष्टतालाई अधिकतम बनाउनु हो।यो धेरै चरहरूको अधिक वा कम अनुमानित प्रभाव द्वारा निर्धारण गरिन्छ।एउटा महत्त्वपूर्ण चर प्राइमरको 3′अन्तमा रहेको अनुक्रम हो।
महत्त्वपूर्ण रूपमा, विशिष्टताको लागि डिजाइन गरिएको PCR assess ले फराकिलो गतिशील दायरामा उच्च दक्षता कायम गर्ने सम्भावना बढी हुन्छ, किनभने परखले गैर-विशिष्ट प्रवर्धन उत्पादनहरू उत्पादन गर्दैन, यसैले PCR अभिकर्मकहरूसँग प्रतिस्पर्धा गर्न वा मुख्य प्रवर्धन प्रतिक्रियालाई रोक्छ।
निस्सन्देह, केहि अवस्थामा, विशिष्टता सबैभन्दा महत्त्वपूर्ण हुँदैन, उदाहरणका लागि, जब लक्ष्य नजिकबाट सम्बन्धित तर विभिन्न रोगजनकहरूको मात्रा निर्धारण गर्ने हो, विशेष डिजाइन, अनुकूलन र प्रमाणीकरण मापदण्डहरू आवश्यक पर्दछ।
पिघलने वक्र एम्प्लिकनहरूको विशिष्टता मूल्याङ्कन गर्नको लागि एक मानक विधि हो, कम्तिमा एकल लक्ष्यलाई विस्तार गर्ने कि नगर्ने सन्दर्भमा।यद्यपि, यो जोड दिनु पर्छ कि पिघलने वक्र भ्रामक हुन सक्छ किनभने, उदाहरणका लागि, तिनीहरू suboptimal प्राइमर र कम टेम्प्लेट सांद्रता को संयुक्त प्रभाव द्वारा प्रभावित हुन सक्छ।
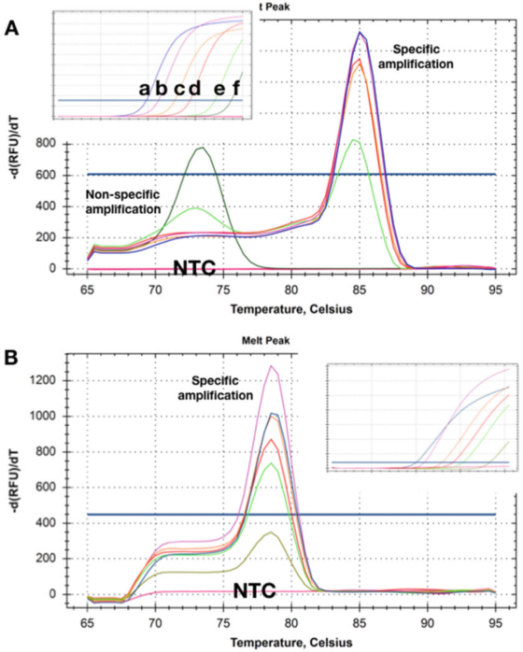
P५ |पग्लने वक्रले दुई लक्ष्य DNA को विभिन्न मात्राको दुई पत्ता लगाउने Tm शिफ्टहरू देखाउँछ।
A. उच्च सांद्रता (ad) मा, qPCR मापन पूरा भएपछि त्यहाँ कुनै स्पष्ट प्राइमर डाइमर छैन।टेम्प्लेट एकाग्रता 50 प्रतिहरू (e) मा घट्दै जाँदा, एक गैर-विशिष्ट उत्पादन देखा पर्न थाल्छ र सबैभन्दा कम एकाग्रता (f) मा मात्र उत्पादन हुन्छ।
B. परीक्षणले सबै लक्ष्य एकाग्रतामा समान Tms रेकर्ड गर्यो, र सबैभन्दा कम एकाग्रता (5 प्रतिलिपिहरू) मा पनि कुनै स्पष्ट प्राइमर डाइमर थिएन।यी दुई पत्ता लगाउने विधिहरू प्रयोग गर्दा, एनटीसीहरूमा कुनै प्रवर्धन उत्पादनहरू फेला परेनन्।
P5 ले नमूनाहरूसँग प्राप्त विघटन वक्रहरू देखाउँछ जसमा टेम्प्लेट विभिन्न सांद्रताहरूमा अवस्थित छ।P 5a ले देखाउँछ कि दुई सबैभन्दा कम सांद्रतामा, उत्पादन गरिएका गैर-विशिष्ट प्रवर्धन उत्पादनहरूको Tms विशिष्ट एम्प्लिकनहरू भन्दा कम छन्।
स्पष्ट रूपमा, यो पत्ता लगाउने विधि कम सांद्रतामा अवस्थित लक्ष्यहरू पत्ता लगाउन विश्वसनीय रूपमा प्रयोग गर्न सकिँदैन।
चाखलाग्दो कुरा के छ भने, NTCs, अर्थात्, कुनै पनि DNA नभएका नमूनाहरूले (गैर-विशिष्ट) एम्प्लीफिकेशन उत्पादनहरू रेकर्ड गरेनन्, यसले पृष्ठभूमि जीनोमिक डीएनए गैर-विशिष्ट प्रवर्धन/पोलिमराइजेशनमा भाग लिन सक्छ भन्ने संकेत गर्दछ।
कहिलेकाहीँ त्यस्ता पृष्ठभूमि प्राइमरहरू र गैर-विशिष्ट प्रवर्द्धनलाई सुधार गर्न सकिँदैन, तर कुनै पनि टेम्प्लेट एकाग्रता र NTC (P 5b) मा गैर-विशिष्ट प्रवर्धन नभएको पत्ता लगाउने विधि डिजाइन गर्न सम्भव छ।
यहाँ, 35 को Cq सँग लक्ष्य एकाग्रताको प्रवर्धन पनि रेकर्ड गर्दा एक विशिष्ट विघटन वक्र उत्पादन हुनेछ।त्यसैगरी, NTCs ले गैर-विशिष्ट प्रवर्धनको कुनै संकेत देखाएन।कहिलेकाहीँ, पत्ता लगाउने व्यवहार आमा रक्सीमा निर्भर हुन सक्छ, र केहि बफर रचनाहरूमा मात्र गैर-विशिष्ट प्रवर्धन पत्ता लगाइन्छ, जुन विभिन्न Mg2+ सांद्रतासँग सम्बन्धित हुन सक्छ।
पत्ता लगाउने स्थिरता
Ta को अनुकूलन qPCR पत्ता लगाउने अनुभवजन्य प्रमाणीकरण र अनुकूलन प्रक्रियामा एक उपयोगी कदम हो।यसले NTC प्रवर्द्धन नगरी न्यूनतम Cq उत्पादन गर्ने तापक्रम (वा तापक्रम दायरा) देखाएर प्राइमर सेटको बलियोपनको प्रत्यक्ष सङ्केत प्रदान गर्दछ।
संवेदनशीलतामा दुई देखि चार-गुणा भिन्नता उच्च mRNA अभिव्यक्ति भएका व्यक्तिहरूको लागि महत्त्वपूर्ण नहुन सक्छ, तर निदान परीक्षणहरूको लागि, यसको अर्थ सकारात्मक र गलत नकारात्मक परिणामहरू बीचको भिन्नता हुन सक्छ।
qPCR प्राइमरहरूको Ta गुणहरू धेरै भिन्न हुन सक्छन्।केही परीक्षणहरू धेरै बलियो हुँदैनन्, र यदि तिनीहरू प्राइमरहरूको इष्टतम Ta मान अन्तर्गत प्रदर्शन गरिएन भने, तिनीहरू चाँडै पतन हुनेछन्।
यो महत्त्वपूर्ण छ किनभने यस प्रकारको पहिचान वास्तविक संसारमा अक्सर समस्याग्रस्त हुन्छ, र नमूनाको शुद्धता, DNA को एकाग्रता, वा अन्य DNA को उपस्थिति इष्टतम नहुन सक्छ।
थप रूपमा, लक्ष्य प्रतिलिपि नम्बर व्यापक दायरामा भिन्न हुन सक्छ, र अभिकर्मकहरू, प्लास्टिकका भाँडाहरू, वा उपकरणहरू परीक्षण सेटअप गर्दा प्रयोग गरिएका भन्दा फरक हुन सक्छन्।
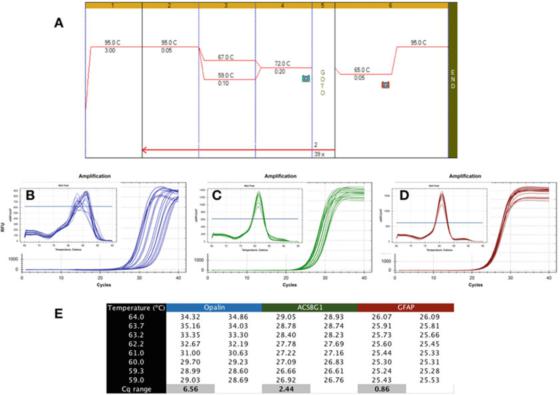
P6|तापमान ढाँचाले PCR पत्ता लगाउने फरक बलियोता देखाउँछ।
A. मानव मस्तिष्क RNA बाट तयार पारिएको cDNA मा PCR प्रदर्शन गर्न Bioline's Sensifast SYBR mastermix (क्याटलग नम्बर BIO-98050) को प्रयोग गर्नुहोस्।
B. Apalene (NM_033207, F: GCCATGGAGGAAAAGTGACAGACC, R: CTCATGTGTGTGGTGATCTCCTAGG) को प्रवर्धन नक्सा र विघटन कर्भ रेकर्ड गर्न Bio-Rad को CFX qPCR उपकरण प्रयोग गर्नुहोस्।
C. ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG) को प्रवर्द्धन ग्राफ र पिघल्ने वक्र।
D. GFAP को प्रवर्द्धन ग्राफ र विघटन वक्र (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCTCGTGGATCTTC)।
E. Cqs 7C तापक्रम ढाँचा अन्तर्गत रेकर्ड गरिएको Cq मा भिन्नता देखाउँदै विभिन्न एनेलिङ तापक्रममा रेकर्ड गरियो।
P 6 ले अवांछनीय परीक्षणको सामान्य नतिजा देखाउँछ, जहाँ qPCR 59C र 67C (P 6a) बीचको ग्रेडियन्ट Tas प्रयोग गरी तीन मानव मस्तिष्क-विशिष्ट जीनको लागि प्राइमरहरू प्रयोग गरी प्रदर्शन गरिएको थियो।
यो एम्प्लीफिकेशन ग्राफबाट देख्न सकिन्छ कि ओपलिन प्राइमरहरू आदर्शबाट टाढा छन् किनभने तिनीहरूको इष्टतम Ta दायरा धेरै साँघुरो छ (चित्र 6b), अर्थात्, Cqs व्यापक रूपमा फैलिएको छ, परिणामस्वरूप Cqs तिनीहरूको इष्टतम Cqs कम तुलनामा उल्लेखनीय रूपमा रहेको छ।
यो पत्ता लगाउने विधि अस्थिर छ र suboptimal प्रवर्धन हुन सक्छ।त्यसैले, प्राइमरहरूको यो जोडीलाई पुन: डिजाइन गर्नुपर्छ।थप रूपमा, पिघलने वक्र विश्लेषण (इनसेट) ले देखाउँछ कि यो पत्ता लगाउने विधिको विशिष्टता पनि समस्याग्रस्त हुन सक्छ, किनभने प्रत्येक Ta को पग्लने वक्र फरक छ।
P 6c मा देखाइएको ACSBG1 पत्ता लगाउने विधि माथिको Opalin पत्ता लगाउने विधि भन्दा बढी बलियो छ, तर यो अझै पनि आदर्शबाट टाढा छ, र यसलाई सुधार गर्न सकिने सम्भावना छ।
यद्यपि, हामी दृढता र विशिष्टता बीच कुनै आवश्यक जडान छैन भन्ने कुरामा जोड दिन्छौं, किनभने यो पत्ता लगाउने विधिद्वारा उत्पादित विघटन वक्रले सबै Tas (इनसेट) मा समान शिखर मान देखाउँछ।
अर्कोतर्फ, P 6d मा देखाइएको GFAP परीक्षणमा जस्तै Tas को विस्तृत दायरामा समान Cqs उत्पादन गर्दै, बलियोपन परीक्षण धेरै सहनशील छ।
एउटै 8 डिग्री सेल्सियस दायरामा प्राप्त Cqs मा भिन्नता 1 भन्दा कम छ, र विघटन वक्र (इनसेट) ले यस तापमान दायरामा पहिचान विशेषताहरू पुष्टि गर्दछ।यो ध्यान दिन लायक छ कि गणना गरिएको Tas र वास्तविक Ta दायरा धेरै फरक हुन सक्छ।
अनुसन्धानकर्ताहरूलाई कुशल प्राइमरहरू डिजाइन गर्न मद्दत गर्न डिजाइन गरिएका धेरै दिशानिर्देशहरू छन्, जसमध्ये धेरैजसो लामो-स्थापित नियमहरूमा आधारित छन् र प्राइमरहरूको 3′छेउमा धेरै ध्यान दिइएको छ।यो प्रायः 3' अन्तमा G वा C र दुई G वा C आधारहरू (GC क्ल्याम्प) समावेश गर्न सिफारिस गरिन्छ, तर अन्तिम 5 आधारहरू मध्ये दुई भन्दा बढी हुँदैन।
व्यवहारमा, यी नियमहरूले अनुसन्धानकर्ताहरूलाई मार्गदर्शन गर्न सक्छन्, तर तिनीहरू सबै परिस्थितिहरूमा सही हुनुपर्छ भन्ने छैन।
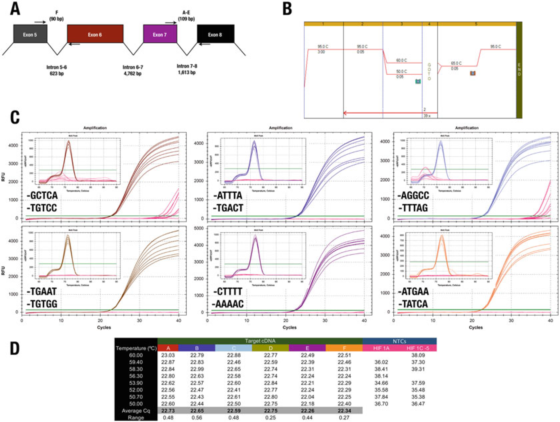
P७ |प्राइमरको 3′छेउले विशिष्टता वा दक्षतामा थोरै प्रभाव पार्छ।
A. मानव HIF-1α (NM_181054.2) जीनको लागि प्राइमरहरूको स्थिति।
B. छवटा परीक्षण वस्तुहरू विस्तार गर्न Agilent Brilliant III SYBR ग्रीन मदर लिकर (Cat. No. 600882) प्रयोग गर्नुहोस्।
C. Bio-Rad को CFX qPCR इन्स्ट्रुमेन्ट र 3′end प्राइमरहरू द्वारा रेकर्ड गरिएको एम्प्लीफिकेशन ग्राफ र पिघल्ने वक्र।NTC लाई रातो रंगमा देखाइएको छ।
D. प्रत्येक परीक्षण वस्तुको Cqs रेकर्ड
उदाहरणका लागि, P 7 मा नतिजाले 3′end नियमको विरोध गर्दछ।सबै डिजाइनहरूले मूलतया समान परिणामहरू उत्पादन गर्छन्, केवल दुई प्राइमर संयोजनहरूले NTC मा गैर-विशिष्ट प्रवर्धनको लागि नेतृत्व गर्दछ।
यद्यपि, हामी GC क्लिपको प्रभावलाई समर्थन गर्न सक्दैनौं, किनभने यस अवस्थामा, A वा T को अधिकतम 30 आधारहरूको रूपमा प्रयोग गर्दा विशिष्टता कम हुँदैन।
परीक्षण C, जहाँ F प्राइमर GGCC मा समाप्त हुन्छ, NTCs मा Cq हरू रेकर्ड गर्यो, जसले 30-अन्तमा यी अनुक्रमहरू बेवास्ता गर्न चाहन्छ भनेर संकेत गर्दछ।हामी जोड दिन्छौं कि प्राइमर जोडीको उत्कृष्ट 3′अन्तको अनुक्रम निर्धारण गर्ने एक मात्र तरिका केही उम्मेद्वार प्राइमरहरू प्रयोगात्मक रूपमा मूल्याङ्कन गर्नु हो।
प्रवर्धन दक्षता
महत्त्वपूर्ण रूपमा, यद्यपि गैर-विशिष्ट PCR पत्ता लगाउने कहिले पनि विशिष्ट हुन सक्दैन, एम्प्लिफिकेशन दक्षतालाई इन्जाइम, मदर लिकर, additives, र साइकल चलाउने अवस्थाहरू परिवर्तन गरेर धेरै फरक तरिकाहरूमा समायोजन र अधिकतम गर्न सकिन्छ।
PCR पत्ता लगाउने क्षमताको मूल्याङ्कन गर्न, लक्ष्य न्यूक्लिक एसिडको 10 वा 5 गुणाको क्रमिक पातलो प्रयोग गर्नु राम्रो हुन्छ, अर्थात् "मानक कर्भ विधि"।
यदि PCR एम्प्लिकन वा सिंथेटिक DNA लक्ष्यहरू मानक वक्र उत्पन्न गर्न प्रयोग गरिन्छ भने, यी लक्ष्यहरूको क्रमिक कमजोरीहरू पृष्ठभूमि DNA (जस्तै जीनोमिक DNA) को स्थिर मात्रामा मिसाइनुपर्छ।
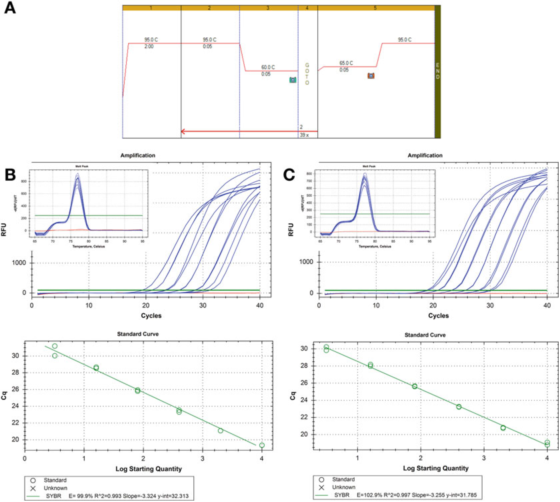
P८ |PCR को प्रभावकारिता मूल्याङ्कन गर्न पातलो कर्भ।
A. HIF-1: F: AAGAACTTTTAGGCCGCTCA र R: TGTCCTGTGTGGTGACTTGTCC र Agilent's Brilliant III SYBR ग्रीन मास्टरमिक्स (क्याटलग नम्बर 600882) PCR र पिघलने वक्र अवस्थाहरूको लागि प्राइमरहरू प्रयोग गर्नुहोस्।
B. 100 ng RNA लाई उल्टो ट्रान्सक्राइब गरिएको थियो, 2 पटक पातलो गरियो, र क्रमिक रूपमा पातलो cDNA नमूनाहरू 5 पटक 1 ng मानव जीनोमिक DNA मा पातलो गरियो।पिघलने वक्र इनसेटमा देखाइएको छ।
C. दोस्रो सीडीएनए नमूनाको लागि आरटी प्रतिक्रिया, कमजोरी, र क्रमिक कमजोरी दोहोर्याइएको थियो, र परिणामहरू समान थिए।
P 8 ले दुईवटा मानक वक्रहरू देखाउँदछ, दुई फरक cDNA नमूनाहरूमा एउटै पत्ता लगाउने विधि प्रयोग गरेर, परिणाम एउटै दक्षता हो, लगभग 100%, र R2 मान पनि उस्तै छ, त्यो हो, प्रयोगात्मक डेटा र रिग्रेसन रेखा वा डेटाको रेखीयता डिग्री बीचको फिटको डिग्री।
दुई मानक वक्र तुलनात्मक छन्, तर बिल्कुल समान छैन।यदि उद्देश्य लक्ष्यको सही मात्रा निर्धारण गर्ने हो भने, यो अनिश्चितताको व्याख्या नगरी प्रतिलिपि नम्बर गणना प्रदान गर्न अस्वीकार्य छ भनेर ध्यान दिनुपर्दछ।
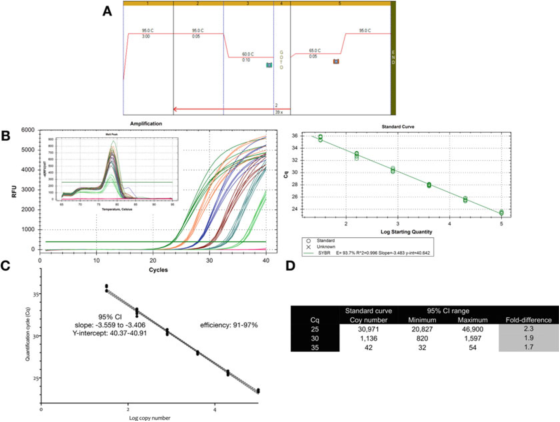
P९ |मानक वक्र प्रयोग गरेर परिमाणीकरणसँग सम्बन्धित मापन अनिश्चितता.
A. GAPDH (NM_002046) को लागि PCR र पग्लने वक्र अवस्थाहरू प्रदर्शन गर्न प्राइमरहरू प्रयोग गर्नुहोस्।F: ACAGTTGCCATGTAGACC र R: TAACTGGTTGAGCACAGG र बायोलाइनको सेन्सिफ्यास्ट SYBR मास्टरमिक्स (क्याटलग नम्बर BIO-98050)।
B. एम्प्लीफिकेशन चार्ट, पग्लने कर्भ र बायो-राडको CFX qPCR उपकरणसँग रेकर्ड गरिएको मानक कर्भ।
C. मानक वक्र ग्राफ र 95% आत्मविश्वास अन्तराल (CI)।
D. dilution curve बाट व्युत्पन्न तीन Cq मानहरूको प्रतिलिपि नम्बर र 95% विश्वास अन्तराल।
P 9 ले देखाउँछ कि एक अनुकूलित परीक्षणको लागि, एकल मानक वक्रको अन्तर्निहित परिवर्तनशीलता लगभग 2 गुणा (95% आत्मविश्वास अन्तराल, न्यूनतम देखि अधिकतम) हो, जुन अपेक्षा गर्न सकिने सबैभन्दा सानो परिवर्तनशीलता हुन सक्छ।
सम्बन्धित उत्पादन:
पशु टिस्यु प्रत्यक्ष पीसीआर किट
पोस्ट समय: सेप्टेम्बर-30-2021